盐藻网摘要
目的:比较扩增盐藻肌动蛋白基因3’旁侧序列的2种PCR方法。方法:用pvuⅡ、EcoRⅤ和StuⅠ3种限制性内切酶消化盐藻基因组DNA后,供2种PCR用,一种是连接介导法PCR(LMPCR),与用人工合成的适配子相连并用适配子引物和盐藻肌动蛋白基因特异引物扩增未知序列;另一种是反向PCR(IPCR),酶切后的DNA自身环化做模板,用2对基因特异引物反向扩增。结果:2轮PCR后,LMPCR中得到大量的非特异扩增产物,测序结果发现许多产物是由适配子引物AP2单独扩增引起。而反向PCR在得到pvuⅡ和StuⅠ消化的自连文库中扩增得到2.5kb特异产物,经测序发现,片段两侧为基因特异引物,部分序列与已知序列相一致。Southern也进一步证实了IPCR扩增片段来源于盐藻基因组DNA。结论:IPCR技术在克隆基因旁侧序列时优于LMPCR方法。
从基因组中获得未知基因序列,传统的方法是标记已知部分序列做探针,然后从该基因组文库中杂交筛选含目的片段的克隆子,再经反复筛选后,进行测序。该方法尽管可靠但操作相当繁琐且效率低下[1]。PCR最初是为扩增已知序列DNA而设计的,而最近一些技术的发展使得未知序列的扩增成为可能,并且已广泛应用[2,3]。作者介绍2种PCR扩增未知序列的方法,并重点探讨2种方法的优劣。一种为反向PCR(inverse PCR,IPCR)[2],即用适宜的限制性内切酶消化基因组DNA,再将消化的产物自身环化,以环化后的片段做模板,用一对与核心区两侧互补的引物做PCR,引物的延伸是从核心区出发,沿环状分子向两侧的未知序列区进行。另一种是连接介导PCR(LMPCR)[3],限制性内切酶消化基因组DNA后,与人工合成的接头相连,然后用2条同向的基因特异引物与接头引物做2次PCR以扩增未知序列。
1 材料与方法
1.1 材料
杜氏盐藻(Dunaliella salina D.S.UTEX1644Teod)购自美国德州大学,TaqDNA聚合酶、连接酶、T-A载体及逆转录酶均为Promega公司产品。Southern杂交试剂盒为PIERCE公司North2SouthHRP直接标记和检测试剂盒。
1.2 方法
参见分子克隆手册。
1.2.1 盐藻细胞的培养
盐藻常规接种到改良的Ben-Amotz盐藻培养液中,置23℃光照培养箱中进行培养。
1.2.2 盐藻细胞基因组DNA的制备
根据CATB方法制备盐藻基因组。
1.2.3 引物
本实验中的引物均用primer primier软件设计,由上海基康生物公司合成。基因特异引物参照D.S.mRNA 3’UTR序列(Genbank accession no.AF163669)。PCR最初是为扩增已知序列DNA而设计的。

1.2.4 连接介导法PCR
用Clontech公司的Genomewalker试剂盒,分别取3种文库(pvuⅡ、EcoRV和StuⅠ)的DNA 1μl为模板,以GSP1和人工合成的接头AP1。采用大连宝生物工程公司的TaKaRa LA Taq DNA polymerase,首轮扩增的参数为,94℃变性30s,68℃延伸4min,6个循环后改为94℃变性30 s,55℃变性1 min,72℃延伸4 min,30个循环终止。将第1次PCR产物稀释10倍,取1μl做第2轮PCR的模板,用基因特异引物GSP2和人工接头引物AP2进行半巢式PCR反应,反应条件同上。取5μl半巢式PCR产物在10 g/L琼脂糖凝胶上进行电泳分离。
1.2.5 反向PCR
参照文献[5],上述4个酶切基因组DNA,分别取10ng,用1μlT4DNA聚合酶,以20μl体积15℃连接过夜。取1μl连接产物作为反向PCR第1轮扩增的模板,引物为GSP1和IGSP1,扩增条件同加接头法PCR,取1μl第1轮PCR产物作第2轮PCR的模板,引物GSP2和IGSP2,反应条件与第1轮相同,取终产物5μl行琼脂糖凝胶电泳。
1.2.6 序列克隆及Southern bloting
将第2轮PCR产物经15g/L琼脂糖凝胶电泳后用胶回收,连接到T-A载体上,转化大肠杆菌JM109细菌,挑选菌落扩增测序。序列翻译与分析用网上软件处理,氨基酸比较用BLAST软件处理。克隆得到的片段用辣根过氧化物酶标记,分别于pvuII、EcoRV和StuI酶切的盐藻核基因组DNA进行杂交,具体步骤按说明书进行。
2 结果
2.1 连接介导法PCR结果
由pvuⅡ、EcoRV和StuⅠ酶切盐藻基因组DNA,纯化后与人工合成的接头相连,构建了3个文库。首先由2个外测引物AP1/GSP1扩增文库,然后以第1次PCR产物为模板,分别用单独的AP1及2个外测引物AP2/GSP2扩增,最后单独用AP2为引物,以AP2/GSP2扩增产物为模板行PCR,电泳结果见图1。3个文库中的PCR结果非特异条带较多,并且在pvuⅡ酶切的文库2 kb附近、EcoRV酶切的文库2.5 kb附近和StuⅠ酶切的文库2 kb附近均在同一个位置上存在扩增产物。
2.2 反向PCR结果
见图2。由图2可知,盐藻基因组DNA经pvuⅡ、EcoRV和StuⅠ酶切再环化后,模板第1次扩增产物均阴性。在第2次PCR中,pvuⅡ和StuⅠ文库在2.5 kb附近出现非常特异的条带,而EcoRV环化文库则呈阴性。
2.3 反向PCR片段Southern bloting分析
为证明从反向PCR片段来源于盐藻基因组DNA,将pvuⅡ环化文库得到的产物用辣根过氧化物酶标记,与pvuⅡ、EcoRV和StuⅠ酶切盐藻基因组产物杂交,均得到特异的杂交信号。
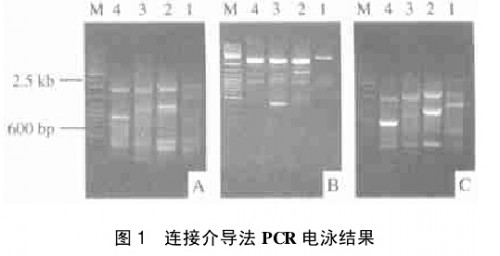
A、B、C分别从pvuÒ、EcoRV和StuÑ文库中2次PCR扩增产物。所应用的引物为:1:AP1/GSP1; 2:AP1; 3:AP2/GSP2; 4:AP2;M:DNA ladder maker
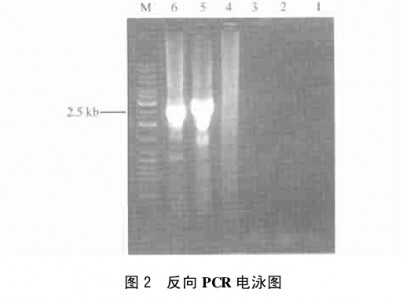
1~3为第1次PCR产物,4~6为第2次PCR产物。DNA环化文库来源:1、4为EcoRV;2、5为StuÑ;3、6为pvuÒ。M:DNA ladder maker
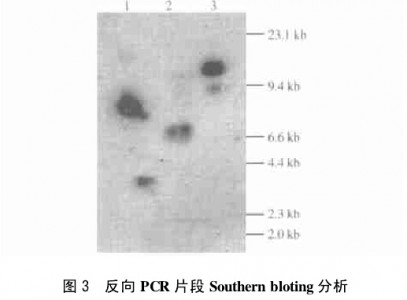
反向PCR片段Southern bloting分析酶切基因组DNA,用pvuII环化文库得到的产物做探针进行消化,基因组DNA所使用的酶,1:pvuÒ;2:EcoRV;3:StuÑ
2.4 序列克隆及测序
将连接介导法PCR和反向PCR结果中2.5 kb附近条带胶回收,克隆到T载体上,大量挑选重组质粒进行测序,发现步行法中插入片段两侧为AP2引物序列,所扩增的产物与已知部分序列不能吻合。反向PCR结果序列则与已知序列相一致。序列结果由Genbank收录(accession no.#43#Journal of Zhengzhou University(Medical Sciences) Jan. 2004 Vol.39 No.1AF539623)。
3 讨论
本研究应用反向PCR法,克隆获得了盐藻肌动蛋白基因3.侧序列,经测序证实与已知盐藻mRNA3.UTR序列部分(Genbank accession no. AF163669)吻合。研究结果显示,本结果中所描述的反向PCR法在扩增基因组未知序列时明显优于连接介导法。连接介导法是比较传统的基因组步行方法(genomic walking),应用加接头法克隆基因组未知序列时,2次PCR结果常常出现假阳性产物,第3次PCR及测序结果均证实扩增产物是由GSP2引物单独反应引起。本研究结果显示,在构建基因组步行方法的文库时,基因组DNA经过平端内切酶切开后,与平端适配子引物相连,理论上,所有两侧为平端的DNA片段都有与适配子连接的机会。所以,无论是第1次PCR还是第2次PCR,都会出现许多由AP1/AP2单引物引起的非特异反应产物,而且这些非特异产物在2次PCR之后均有所加强。因此,大量非特异产物蒙蔽了阳性结果,造成/特异0假象,同时给科研工作造成不必要的浪费。本实验研究证实,反向PCR法克服了上述缺点:¹运用4条特异引物,再经过2次PCR,大大提高了产物的特异性。º操作简便、快捷。即克服了文库筛选中克隆、亚克隆的繁琐步骤,也避免了连接介导法文库构建中存在的一些问题。»可以扩增任何一个已知基因片段两侧未知序列,而连接介导法仅能扩增单侧序列。本课题组主要从事转基因盐藻研究,其中在建立盐藻遗传背景时克隆得到了许多转基因必需的基因片段(Gen-Bank accession AF541981,AY232669,AF541875),这些片段主要通过反向PCR获得,并在转基因体系建立中发挥了重要作用。
本实验中反向PCR的模板,是由3个内切酶充分消化基因组DNA而产生的,通常选择已知序列中不存在的、又较常见的6个碱基组成的内切酶来酶切基因组DNA。此外,酶切后模板DNA的浓度很重要,本实验中使用了2 mg/L,如果高于这个浓度,可能会形成片段之间的连接,以致于无法得到特异片段。
致谢 本工作在河南省分子医学重点学科开放实验室完成。
全文下载>>>2种PCR方法扩增盐藻肌动蛋白基因<<<解压密码:www.yanzao360.com
原文链接:http://www.yanzao360.com/695.html








