自从Teodoresco1905年对不同生境的盐藻(Dunaliel-la.salina)进行比较以来,已有29个种的盐藻种类被分类鉴定。传统的盐藻分类常基于细胞学以及生理特征,其中,细胞学特征包括细胞的形态(包括大小、形状和对称性)、叶绿体的形状及淀粉核、眼点和细胞质内颗粒的有无等。盐度的适应性以及β类胡萝卜素的积累能力等常是盐藻分类的重要生理生化指标。但是这些细胞学及生理学的指标很大程度上受到环境因素的影响,缺乏统一的标准,因而盐藻的系统分类仍相当混乱,给相关研究带来很大困难。
Borowitzka和Siva[1]曾对杜氏盐藻的传统分类进行过详细的评述,并对22个杜氏盐藻种重新进行了修订,同时也对盐藻基因的分子特征用于盐藻种类鉴定的优势进行了评价,认为分子特征不受环境因素的影响,可作为传统分类系统的有效补充。核糖体5.8SrDNA及其与大亚单位28S rDNA和小亚单位18S rD-NA临近的2个间隔区序列ITS(分别称为ITS1和ITS2)具有相当低的遗传保守性,是进行生物种内及种间鉴别的理想分子标记,Mariela等曾利用这一序列的多态性对多个盐藻种的亲缘关系进行过研究,王伟等曾根据ITS的序列多样性对两株盐藻进行了种类鉴定。
本实验室对保存在藻种库的7株盐藻细胞学及盐度适应性的初步研究表明,这7株微藻的细胞学特征极为相似,均含有两根鞭毛,细胞形状呈长椭圆形或梨形,眼点不明显,培养至指数增长期所有培养液均呈浓绿色。在高盐度条件下7株盐藻甘油合成能力极剧增强,而β胡萝卜素的含量增加并不明显,这与描述的绿色杜氏藻的特征极为相似,为便于对这些藻株的研究利用,有必要借助分子生物学技术对这7株盐藻与其它盐藻种的亲缘关系进行分析鉴定。
作者简介:姜 玮(1985-),女,硕士生,研究方向:微藻生理和生态学。中国海洋大学学报
1 材料与方法
1.1材料
本实验涉及的7株盐藻,均取自中国海洋大学微藻种质库,经单克隆纯化后,在盐度为100的f/2培养基中丰富培养。
1.2基因组DNA提取
采用改良的CTAB法提取盐藻的DNA,具体操作为,离心收集对数生长期的盐藻细胞,每500LL浓缩藻液加入500LL 65e预热的CTAB提取缓冲液,65e温育1 h后,等体积Tris饱和酚抽提1次,酚-氯仿-异戊醇重复抽提2次。离心,上清液转至新的离心管中,加入2倍体积的无水乙醇,于4e静置沉淀2 h。沉淀用70%乙醇洗涤2次,自然干燥后,溶于50LL去离子水中备用。
1.3 5.8S rDNA及ITS1和ITS2序列扩增PCR扩增
7株盐藻5.8S rDNA及ITS1和ITS2序列,正向扩增的引物序列ITSA为: 5.-GGAAGGA-GAAGTCGTAACAAGG-3.,反向扩增的引物序列ITSB为: 5.-TCCTCCCTTATTGATATGC-3.[3]。扩增引物由上海生工生物工程技术服务有限公司合成。
PCR反应体系(50LL)为,2LL模板DNA,0.2Lmol/L扩增引物,0.1Lmol/L dNTPs,1.5 mmol/L Mg2+,3U
Taq酶和1倍反应缓冲液。PCR反应条件为,94e延伸5 min,随后94e40 s、53e40 s、72e1 min,进行30个循环,并在72e延伸10 min。扩增产物经1.5%低熔点琼脂糖凝胶电泳分析,收集1 000 bp左右扩增序列,纯化后直接交由上海生工生物工程技术服务有限公司完成测序。
测回序列采用DNAstar软件进行序列拼接和比对,经在NCBI服务器(http://www.ncbi.nlm.nih.gov/)上用BLAST软件进行同源性检测,以确证扩增序列。扩增序列与Genbank搜索的27个盐藻,3个衣藻的相应序列(见表1)用Clustalx进行多序列匹配排列和比较,采用MEGA4.0构建进化树,自举数据集1 000次作置信度检测。
1.4 RAPD分析
以上海生工生物工程技术服务有限公司合成的30对10碱基序列作为RAPD分析的随机引物,进行PCR扩增,20LL PCR扩增体系含有,dd H2O 12.9LL,10@PCR Buffer 2LL,dNTPs(2.5 mmol/L) 1.5LL,Mg2+(20 mmol/L)1.6LL,随机引物(1Lmol/L)1LL,模板DNA(50 ng/LL)1LL,Taq酶(2.5 U/LL)0.4LL。PCR扩增条件为,在94e延伸5 min,随后94e1min、36e1 min、72e2 min,进行45个循环,在72e延伸7 min。将扩增产物在含有EB的1.5%琼脂糖凝胶中电泳,以2次RAPD扩增重复中清晰并稳定出现的扩增带作为统计对象,有电泳条带记为1,无电泳带记为0。POPGEN32软件计算多态位点百分率,遗传距离及聚类分析。
2 结果分析
2.1根据5.8S rDNA和ITS序列进行亲缘关系分析
以7株微藻提取的DNA为模版,PCR扩增核糖体5.8S rDNA和ITS序列,结果7株盐藻均扩增到序列长度在600 bp以上,测定序列拼接后在Genbank进行Blast搜索,结果表明,7株盐藻的扩增序列与盐藻的5.8S rDNA和ITS序列的相似性均在85%以上。据此可以确证本研究扩增的序列为扩增的目的序列。
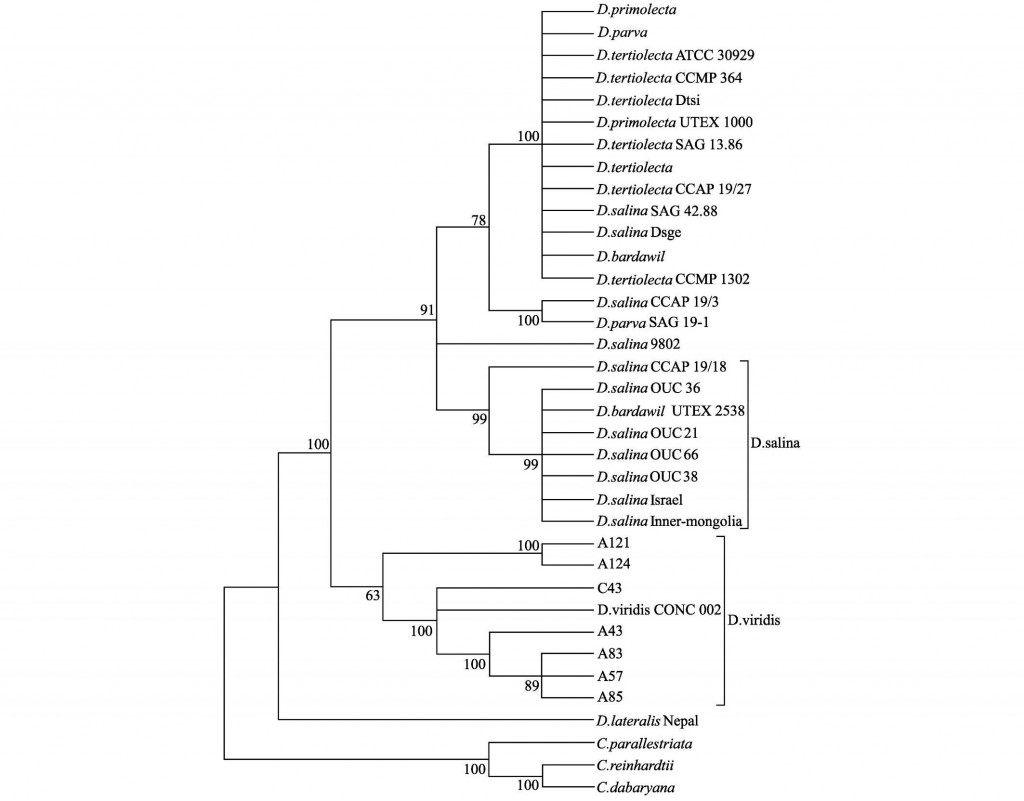
图1 基于7株盐藻的扩增序列和Genebank搜集的27个杜氏藻及3个衣藻的5.8S rDNA和2个ITS区序列构建的UPGMA树
以3个衣藻种作为外类群,将基因库搜索的27个杜氏盐藻5.8S rDNA和ITS序列信息与本研究7株盐藻扩增的序列同时用于亲缘关系分析,采用Meg4.0软件构建系统进化树,分析结果表明构建MP树、NJ树和UPGMA树基本一致,从构建的进化树可以看到(见图1),34个盐藻分别聚为3个大群,7个D.tertiolecta品系与2个D.salina品系(品系代号分别为SAG42.88和DsGe)、2个D.primolecta品系(其中1个代号为U-TEX1000)、1个D.parva品系和1个D.bardawil品系(品系代号为Y4)完全聚为1支,随后与D.salina品系CCAP19/3和D.parva品系SAG19-1聚为的1支聚为1个大支,7个D.salina品系与1个D.bardawil品系(品系代号为UTEX2538)聚为1个大支,D. salina品系9802位于两大支之间。本研究7株盐藻与D.viridis品系CONC 002聚为1个大支,而D.lateralis品系Nepal则单独分支,与其它盐藻种的亲缘关系均较远,结果说明本研究的7株盐藻均为D. viridis。
2.2 7株盐藻基因组DNA的多态性
从上图的进化树还可以看到,7株盐藻亲缘关系也存在一定的差异,其中A57、A83和A85首先聚为1支,随后与A43和C43依次相聚,A121和A124则与前述5株盐藻的亲缘关系较远,为进一步比较7株盐藻的亲缘关系,本研究还对7株盐藻进行了RAPD分析。
利用从30对引物所筛选出的12个10 bp随机引物对7株盐藻进行PCR扩增,每个引物重复2次,选取重复性好的DNA扩增谱带用于统计分析。结果表明,扩增序列的片段大小在250~1 500 bp之间,每个引物扩增带数6~8条不等,平均每个引物扩增带数为7.08条,多态性条带为5.5条。其中,引物S11扩增出的多态性条带百分率最高,为100.00%,引物S17最低,为57.14%。12个引物共扩增出85条谱带清晰重复性好的DNA带,其中66条具有多态性,占总带数的77.65%,显示丰富的多态性。
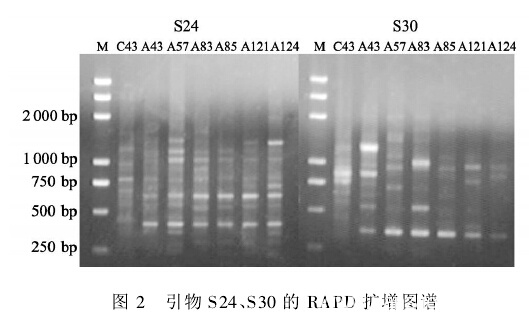
Fig.2 RAPD profiles generated by primers S24 and S30
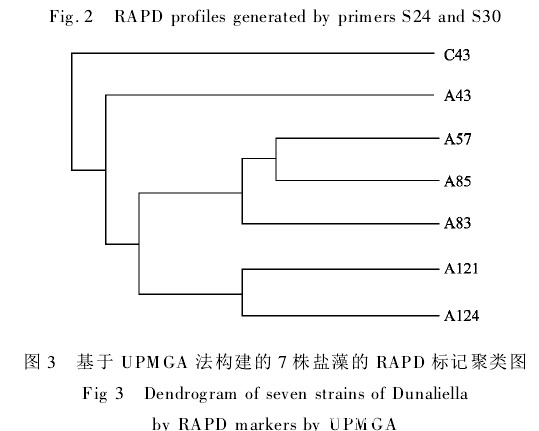
由扩增条带的多态性计算遗传距离(见表3),结果表明,A57和A85的遗传距离最小为0.200 7,最大距离在C43与A83和A85之间,均为0.788 5。从聚类图可以看到A85和A57首先聚为1支,再与A83聚为1大组,A121和A124聚为1组,C43和A43则分别单独聚为1组。这一结果与核糖体5.8S rDNA和ITS序列分析的结果基本相似,不同之处在于RAPD分析结果似乎C43和A43与A57、A85和A83的距离较A121和A124远,这种差异可能与7株藻自身的遗传差异本来就小以及分析时分析单元数目不同有关。
3 讨论
Olmos等采用PCR-RFLP技术对D. parva,D.salina, D.tertiolecta和D.bardawil4个杜氏盐藻核糖体18S rDNA序列进行了分析,根据18S rDNA可变区的长度多态性,可将D.salina, D. parva和D.bardawill分开,但未将D.salina和D. tertiolecta清楚分开。Mariela等[2]采用PCR-RFLP技术对7个杜氏盐藻种(D.salina, D.bardawil, D.tertiolecta, D.parva, D.viridis, D. lateralis, D.peircei)共12个品系核糖体5.8S rDNA和2个ITS序列的长度多态性进行了比较,结果2个D.tertiolecta品系和D.parva及D.peircei各1个品系完全聚为1组,D.salina的1个品系与D.bardawil的1个品系及1个D.viridis品系与D.parva的1个品系也分别优先聚为1支,D.lateralis与其它杜氏盐藻的亲缘关系均较远。孙国华等[5]利用RF-RAPD技术研究了7个已定种的杜氏盐藻的分子特征,并对3个未定种进行了鉴定,根据RAPD结果构建UPGMA树和N-J树,结果3个D.salina品系(分别为D.salinaOUC2、OUC3和OUC4)优先聚类,另一个D.slina品系(D.salinaOUC1)则
与D.bardawil聚为一组,D.parva和D.primolecta聚为一组。张会永等采用RAPD技术对7个杜氏盐藻种(D.salina, D.bardawil, D.minuta, D.biocu-ata, D.parva, D.peircei和D.primolecta)分析结果表明,D. salina, D. bardawil和D. minuta聚为一类,而D.bioculata, D. parva, D. peircei和D. pri-molecta聚为另一类,认为这7个杜氏盐藻种分为2个种比较合适。与长度多态性分析相比,直接进行碱基序列比对获得的亲缘关系应更为可靠,目前,提交至Genbank的杜氏盐藻5.8S rDNA和2个ITS序列已相当丰富,许多已明确种类的杜氏盐藻5.8S rDNA和2个ITS序列信息被完成测序,多个研究者对多个株系的盐藻进行测序,这使比较多个盐藻种的亲缘关系成为可能。本研究将从基因库中分属于8个盐藻种的27条序列信息与本研究扩增的7个未定名盐藻的5.8SrDNA和2个ITS序列同时用于比较分析,结果发现,7个D.tertiolecta品系与2个D.primolecta品系、2个D.salina品系和1个D.parva品系完全聚为1支,而6个盐生杜氏藻(D.salina)品系与1个D.bardawil品系优先聚为1支。这些结果说明利用分子标记进行杜氏藻的亲缘关系分析尽管与传统的分类结果大致一致,但确实还存在明显的不同,这正像Borowitzka和Siva等指出的那样,在当前缺乏同一标准的情况下,分子标记只能作为种类鉴定的辅助手段。本研究7株未定种盐藻与绿色杜氏藻(D.viridis)关系最为紧密,可以进一步证实我们根据细胞学及生理生化分析对7株盐藻的鉴定结果,说明这些微藻均是绿色杜氏藻的不同株系。
根据5.8S rDNA和ITS序列进行7株微藻亲缘关系的分析结果,7株微藻可以分为4组,其中A57,A85和A83为1组,A121和A124为1组,A43和C43分别为另外2组,这一结果与本研究进行RAPD分析的结果极为相似。作者曾对这7株盐藻的生长潜力、盐度适应性、温度适应性和光照强度适应性进行了比较,结果发现,7株微藻对温度和光照强度的适应性相似,而对盐度的适应性差别相当大,如A85在优化的生长条件下相对生长速率最高,A57对高盐度的耐受性最强,而A85在高盐度下4 h内即全部死亡。但5.8S rDNA和ITS序列以及RAPD分析的果并未显示其遗传差367增刊姜 玮,等:7株盐藻的5.8S rDNA和ITS序列及RAPD分析异与盐度适应性存在直接的连锁关系,也许该分子标记方法不适合进行微藻盐度适应性遗传连锁关系的研究。有关这7株盐藻盐度适应性的遗传背景还有待进一步研究。
全文下载>>>7株盐藻的5_8SrDNA和ITS序列及RAPD分析_姜玮<<<解压密码:www.yanzao360.com
原文链接:http://www.yanzao360.com/723.html





